U-Net: Convolutional Networks for Biomedical Image Segmentation
https://link.springer.com/chapter/10.1007/978-3-319-24574-4_28
Abstract
Introduction
Network Architecture
Training
Data Augmentation
Experiments
Conclusion
Abstract
data augmentation의 효율적 활용을 위한 네트워크 훈련 전략 제시
- context 파악을 위한 contracting path
- 정밀한 위치 지정을 위한 symmentric expanding path
→ 아주 적은 이미지로도 end-to-end 훈련이 가능하도록 함
이전 SoTA인 sliding window convolution network 능가
빠른 속도 & 높은 정확도 달성
Introduction
computer vision 분야
한동안의 state of art : DNN
이후 사용 가능한 training dataset과 네트워크 크기 문제로 정체되었다가 ImageNet의 백만개 훈련 이미지를 활용하여 수백만 매개변수를 가진 8층 네트워크가 학습되면서 더 크고 깊은 네트워크들의 학습이 시작됨
이후 convolution neural network의 경우,
classification에서 하나의 이미지 ⇒ 하나의 class label만 사용하는 것이 한계로 발동됨
- 특히 biomedical 분야, 원하는 출력은 location이 필수
- location이 가능하기 위해서는 각 픽셀에 class가 label되어야함
- 특히 biomedical 분야는 수천개의 학습 데이터를 구하기 힘듦
⇒ 💡 Sliding-window
픽셀 주변의 patch를 입력으로 제공하여 각 픽셀의 class label 제공
- 위치 location 가능
- patch 측면으로 훈련하여 데이터의 수 증가
- 네트워크가 각 패치마다 별도로 실행되어 매우 느린 속도이며 겹치는 패치가 많아 중복 발생
- 위치 정확도 ↔ 맥락 상충 발생
- 큰 patch : 위치 정확도를 떨어뜨리는 max-pooling 필요
- 작은 patch : context 정보 감소
본 논문의 네트워크
fully convolutional network 기반
→ 매우 적은 수의 training image만으로 더 정밀한 segmentation 가능
idea) 보통의 축소 경로:contracting path에 pooling 대신 up-sampling 적용
- 높은 해상도
- 정밀한 위치 지정 가능
- 축소 경로의 고해상도 특징들을 up sample된 출력과 결합
- 정밀한 위치 지정 가능
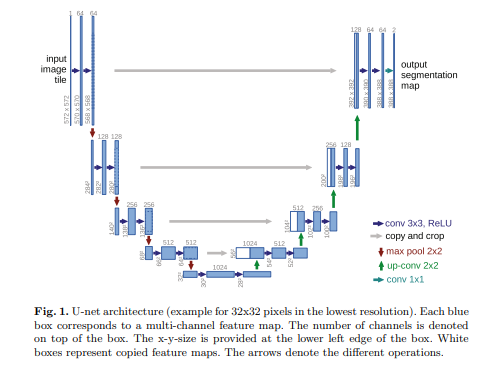
up-sampling 적용하여 고해상도의 출력을 얻음
이때도 많은 수의 feature channel을 가지고 있어 context 정보를 더 높은 해상도 layer로 전파할 수 있게 함
- expanding path는 contracting path와 대체로 대칭을 이룸
- U자 형태 architecture
- fully connected layer 존재하지 않고 합성곱의 유효한 부분만 사용
- segmentation map의 경우, 전체 이미지에서 전체 맥락이 사용가능한 픽셀만을 포함하여 적용
→ overlap-tile strategy
임의의 크기 이미지를 끊임없이 분할 가능
이미지의 경계 픽셀 예측을 위해 누락된 맥락은 입력 이미지를 미러링하여 외삽
but, GPU의 해상도 제한 가능성 → 큰 이미지에 적용해야함
- 적은 수의 훈련 데이터이기 때문에 elastic deformation을 적용하여 data augmentation을 적용함
- 현실적인 변형을 효율적으로 시뮬레이션 적용 가능
elastic deformation
elastic deformation
탄성체의 특성을 이용하여 변형을 줌
탄성이란 연속체에서 어떠한 힘이나 시간의 흐름으로 인해 변화가 발생할 때,
이 힘이 제거된 후 다시 원래대로 돌아오는 것

Network Architecture

[contracting path] [expanding path]
-> contracting path의 경우 전형적인 합성곱 네트워크 구조이다
-> expanding path의 경우 up-sampling 적용하며 축소 경로의 해당 크롭 피쳐맵과 연결됨
각 피쳐맵은 ReLU가 적용되며 모든 합성곱은 경계 픽셀의 손실로 인해 crop을 적용함
최종 layer의 경우, 각 벡터를 원하는 클래스 수로 매칭하기 위해 1*1 합성곱이 적용됨
Training
input image와 해당 segmentation map을 stocastic gradient descent를 사용하여 네트워크 훈련
padding 되지 않은 합성곱 신경망 적용으로 인해 output 이미지는 input 이미지보다 작아짐
overhead 방지와 GPU 최대 사용을 위해 큰 배치크기를 적용하지 않았고, 큰 입력 타일을 적용함
에너지 함수는 최종 피쳐맵 위 픽셀별 softmax와 cross entropy loss function과 결합하여 계산됨
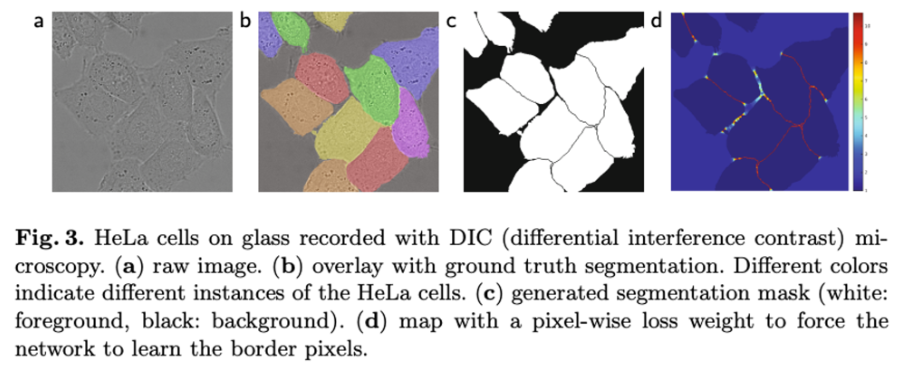
이때, 학습 데이터셋에서 특정 클래스의 픽셀 빈도가 다른 것을 보상하기 위해
segmentation의 groundtruth에 대한 weight map을 사전에 계산 → 우리가 도입한 작은 분리 경계 학습
Data Augmentation
현미경 이미지의 경우, 이동 및 회전 불변성 뿐만 아니라 변형과 회색 값 변화에 대한 견고성 필요
그 중 훈련 샘플의 무작위 탄성 변형은 매우 적은 수의 어노테이션된 이미지로 segmentation network를 훈련시키는 핵심 개념 elastic deformation
- 3*3 그리드에서 random displacement vectors를 사용해 부드러운 변형 생성
- 이때 변위는 10 픽셀 표준 편차를 가진 가우시안 분포에서 샘플링되며 픽셀별 변위는 바이큐빅 보간을 사용해 계산됨
- 축소 경로 끝의 drop out layer는 추가적인 암시적 데이터 증강을 수행함
Experiments
3가지 다른 segmentation 작업에 대한 u-net 적용
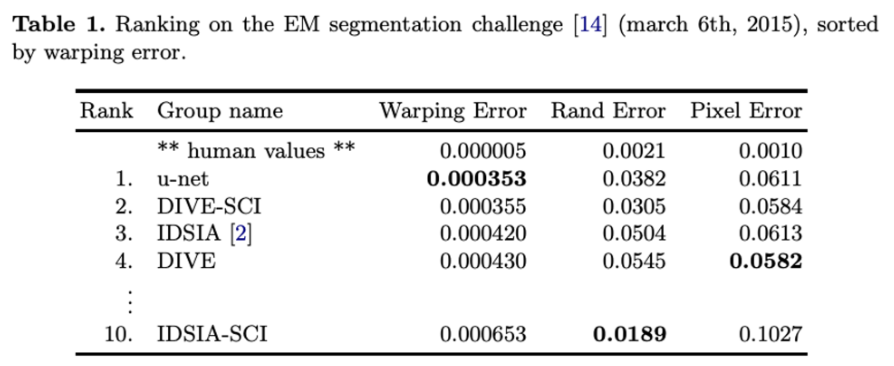
1) 전자 현미경 기록에서의 신경 구조 분할

초파리 첫 번째 유충의 배신경삭의 연속 단면 전송 전자 현미경으로부터 얻은 30개 이미지 세트
각 이미지는 흰색:세포 / 검은색:막으로 완전히 어노테이션된 grountruth segmentation map과 함께 제공
테스트 셋의 분할 맵은 비밀, 예측된 막 확률 맵 평가 → 임계 값에 대한 여러 값 계산
입력 데이터의 7가지 회전 버전을 평균낸 결과로 어떠한 추가적인 사전 처리나 사후 처리 없이도 최고 점수기록, 슬라이딩 윈도우 합성곱 결과보다 상당히 좋음을 보임
2) 광학 현미경 이미지에서의 세포 분할 작업 : PhC-U373
위상 대비 현미경으로 기록된 데이터로, 35개 부분적 어노테이션된 training image를 포함하여 평균 92%를 달성하며 월등한 성과 도출
3) 광학 현미경 이미지에서의 세포 분할 작업 : DIC-HeLa
평평한 유리 위 HeLa 세포를 차등 간섭 대비 현미경으로 기록한 것으로 20개 부분적 어노테이션된 훈련 이미지를 포함하여 이 또한 좋은 성능을 도출
⇒ 3가지 작업에 대한 u-net의 적용은 네트워크가 어노테이션된 훈련 데이터가 매우 제한적임에도 다양한 분야에서 우수한 분할 성능을 제공함을 알 수 있
Conclusion
매우 다양한 biomedical segmentation 응용 프로그램에서 좋은 성능을 달성
- 탄성 변형을 이용한 데이터 증강 → 매우 적은 수의 어노테이션된 이미지
- 단 10시간의 합리적 훈련 시간
'Paper Review > Computer Vision' 카테고리의 다른 글
| CAM : learning deep feature for discriminative localization (0) | 2024.01.27 |
|---|
